Genetic Structure of Wild Bonobo Populations: Diversity of Mitochondrial DNA and Geographical Distribution川本芳,
竹元博幸, 樋口翔子, 坂巻哲也, John A Hart, Terese B Hart,
徳山奈帆子, Gay E Reinartz, Patrick Guislain, Jef Dupain, Amy K
Cobden, Mbangi N Mulavwa, Kumugo Yangozene, Serge Darroze, Celine Devos,
古市剛史 ボノボ(Pan paniscus)はコンゴ川の南部支流域をすべて含む地域に生息しています。ボノボ個体群の遺伝的多様性と進化的関係を調査することを目的に、種生息域の東西両端を含む7個体群(Malebo、Lac Tumba、Lomako、Salonga、Wamba、Iyondji、TL2)を対象に、376の糞試料からミトコンドリアDNA(mtDNA)非コード領域の全塩基配列を決定しました。試料として重複がなく比較可能な136検体(個体群当たり7~37検体)中に、54種類のハプロタイプが区別できました。これらはA1、A2、B1、B2、C、D、の6つの分子系統(クレード)に分けられました。このうちDクレードは今回初めて発見したものです。mtDNAハプロタイプには地域局在性が認められ、45タイプ(全タイプの83%)がひとつの個体群だけに存在していました。このため、生息域中のタイプ分布や個体群内の遺伝的多様性には強い地域差が認められました。調べた7個体群は遺伝距離から東部、中央部、西部の3集団(コホート)に分類できました。地質学的状況の時間的変化をさらに調べる必要はありますが、遺伝的多様性の地理的パターンは更新世の環境変動により形成されてきたと考えられます。TL2を他所から分断するロマミ川は例外として、現存する個体群間の遺伝子流動に関しては、従来主張されてきたような河川障壁の影響はあまり認められませんでした。中央部の集団は高い遺伝的多様性を維持しており、特異なハプロタイプで構成されるCクレードとDクレードは、それぞれ中央部集団のWamba/Iyondji個体群と、東部集団のTL2個体群だけに観察できました。また、他地域から孤立しているMalebo、Wamba、TL2の個体群では、他の個体群に比べて遺伝的多様性が低くなっていました。これらの知見はボノボの保全計画に寄与するものと考えます。
掲載日 3月27日
Website http://www.plosone.org/article/info:doi/10.1371/journal.pone.0059660
(注)
本研究は平成22~24年度環境研究総合推進費(環境省委託事業)の援助により行った国際共同研究です。 PLoS ONE 8(3): e59660. doi:10.1371/journal.pone.0059660 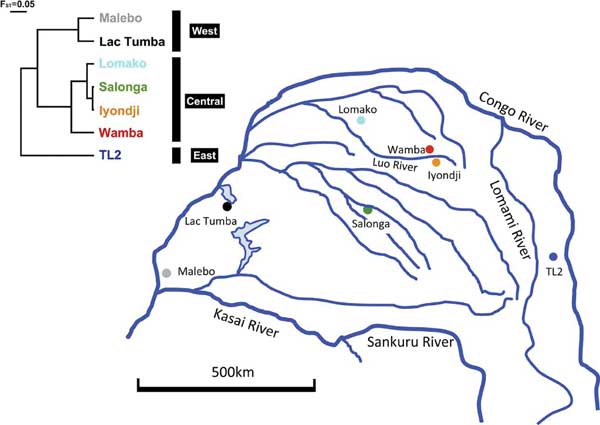
調査個体群の生息地と遺伝的分化
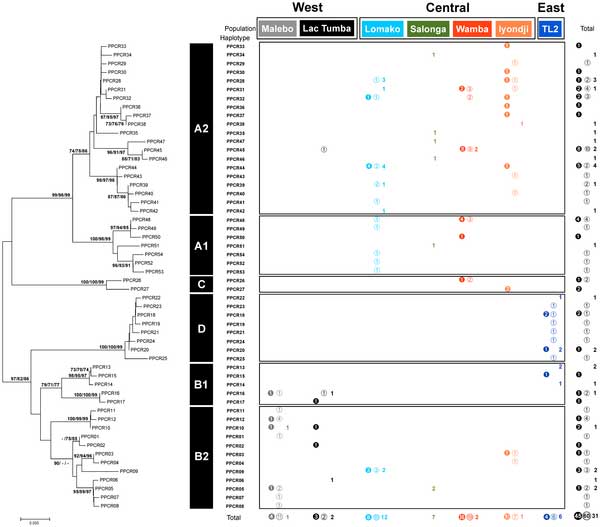
ミトコンドリアDNAタイプの系統関係と地理的分布
Bonobos
(Pan paniscus) inhabit regions south of the Congo River including all areas between its southerly tributaries. To investigate the genetic diversity and evolutionary relationship among bonobo populations, we sequenced mitochondrial DNA from 376 fecal samples collected in seven study populations located within the eastern and western limits of the species’ range. In 136 effective samples from different individuals (range: 7–37 per population), we distinguished 54 haplotypes in six clades (A1, A2, B1, B2, C, D), which included a newly identified clade (D). MtDNA haplotypes were regionally clustered; 83 percent of haplotypes were locality-specific. The distribution of haplotypes across populations and the genetic diversity within populations thus showed highly geographical patterns. Using population distance measures, seven populations were categorized in three clusters: the east, central, and west cohorts. Although further elucidation of historical changes in the geological setting is required, the geographical patterns of genetic diversity seem to be shaped by paleoenvironmental changes during the Pleistocene. The present day riverine barriers appeared to have a weak effect on gene flow among populations, except for the Lomami River, which separates the TL2 population from the others. The central cohort preserves a high genetic diversity, and two unique clades of haplotypes were found in the Wamba/Iyondji populations in the central cohort and in the TL2 population in the eastern cohort respectively. This knowledge may contribute to the planning of bonobo conservation. APR/3/2013
Copyright(C) 2012 PRI ( ). ).
|







