A novel composite retrotransposon derived from or generated independently of the SVA (SINE/VNTR/Alu) transposon has undergone proliferation in gibbon genomes原暢・平井百合子・スダラ バイシャルン・早川卓志・平井啓久・古賀章彦 【背景】3種類の因子が融合して生じたと考えられるレトロトランスポゾン(RNA を中間体として転移するトランスポゾン)がある。3種類の因子は SINE (short interspersed element)、VNTR (variable number of tandem repeat) sequence,
Alu であり、このため複合型レトロトランスポゾンはSVA 因子とよばれている。ヒト上科の種に広く分布し、ヒト上科を除く霊長類にはみられない。SVA 因子の生成の機構は不明である。
【目的】3種類の構成因子に関して、これでなければならないという必然性は、いまのところ認められていない。もし必然性がないのであれば、SVA 因子と同様の機構で生じた他の複合型レトロトランスポゾンが存在することが、可能性として考えられる。あると仮定してそれを同定することを目指した。
【結果】公共のゲノムデータベースが整備されているヒト上科の種について、データベースの検索を行った。その結果、ホオジロテナガザル
(Nomascus leucogenys) で、SVA 因子と類似の因子がみつかった。SINE の部分がなく、そこに
prostaglandin reductase 2 (PTGR2) 遺伝子の一部を含むものであった。そこでこの因子を PVA 因子と名付けた。染色体標本へのハイブリダイゼーションを行い、PVA 因子が散在型反復配列として染色体上に存在することを確認した。また、ゲノムDNAへのサザハイブリダイゼーションから、テナガザル科の4属はすべて PVA 因子をもつこと、およびヒト科にはみられないことが判明した。
【意義】 SVA 因子 の構成要素である SINE は、それ自体が小型のトランスポゾンである。これに対して、相当する位置にPVA 因子にあるPTGR2 遺伝子は、核内のシングルコピーの遺伝子である。このことから、この位置に収まる要素がトランスポゾンでなくても複合型レトロトランスポゾンの形成は起こり得ることといえる。PTGR2 遺伝子である必然性はいまのところみられず、形成の条件はこれまでに考えられていたより緩いものであることが示唆される。 Genes & Genetic Systems 87 (3): 181-190. The superfamily Hominoidea (hominoids) comprises two families: Hominidae (hominids) and Hylobatidae (gibbons, also called small apes). The SVA transposon is a composite retrotransposon that occurs widely in hominoids and is considered to have been generated by stepwise fusions of three genetic elements: SINE-R, a variable number of tandem repeat (VNTR) sequence, and
Alu. We identified a novel transposon whose basic structure is the same as that of SVA, with one prominent difference being the presence of part of
prostaglandin reductase 2 (PTGR2) in place of SINE-R. We designate this composite transposon as PVA and propose two possible mechanisms regarding its generation. One is the derivation of PVA from SVA: the SINE-R region of SVA was replaced with
PTGR2 fragments by template switching. The other is the formation of PVA independently of SVA: a
PTGR2 fragment was fused to an evolutionary intermediate comprising the VNTR and
Alu regions. The nucleotide sequence of the junction between the VNTR and
PTGR2 regions supports the second hypothesis. We identified PVA in the white-cheeked gibbon Nomascus leucogenys by analysis of genome sequence databases, and subsequent experimental analysis revealed its presence in all four gibbon genera. The white-cheeked gibbon harbors at least 93 PVA copies in its haploid genome. Another SVA-like composite transposon carrying parts of the LINE1 and Alu transposons in place of SINE-R, designated as LAVA, has recently been reported. The significance of the discovery of PVA is that its substituted fragment originates not from a transposon but from a single-copy gene. PVA should provide additional insights into the transposition mechanism of this type of composite transposon; the transposition activity is conferred even if the substituted fragment is not related to a
transposon. 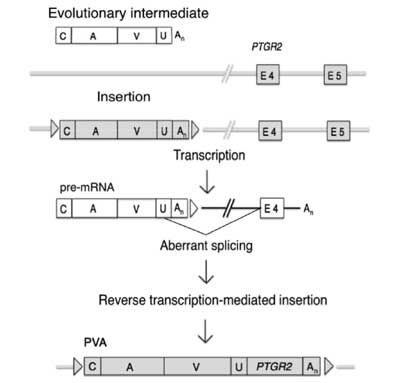 AUG/17/2012
Copyright(C) 2012 PRI ( ). ).
| 






